Necessites ajuda?
Posa't en contacte amb nosaltres

Necessites ajuda?
Posa't en contacte amb nosaltres

Gràcies per contactar
El teu formulari s'ha enviat correctament. El nostre equip es posarà de nou en contacte amb tu com més prompte millor.
Upppss...!! Ha ocorregut un error.
Intente l'enviament més tard o escriga un correu electrònic directament a areaempresas@ua.es

 TECNOLOGIA PATENTADA
TECNOLOGIA PATENTADA
LA FITXA
COMPLETA
BAIXA
RESUM
EXECUTIU
DADES DE CONTACTE:
Relaciones con la Empresa
Oficina de Transferencia de Resultados de la Investigación-OTRI
Universidad de Alicante
Tel.: +34 96 590 99 59
Email: areaempresas@ua.es
http://innoua.ua.es
El grupo de Microbiología Molecular ha desarrollado una herramienta CRISPR, basada en una nueva proteína Cas9, que resuelve algunas limitaciones de las herramientas CRISPR-Cas9 utilizadas habitualmente.
La tecnología tiene aplicación en ingeniería genética y producción de antibacterianos, en los sectores biomédico, agroalimentario y biotecnológico.
Se buscan entidades interesadas en la adquisición de la tecnología para su explotación comercial o para el desarrollo de nuevas aplicaciones o herramientas.

Las regiones CRISPR (por el inglés Clustered Regularly Interspaced Short Palindromic Repeats) del genoma de bacterias y arqueas están constituidas por un número variable de repeticiones cortas (23-48 pb) de ADN, que son las que dan nombre al acrónimo, intercaladas por secuencias no redundantes (denominadas espaciadores) de tamaño semejante al de la repetición. Cada una de las moléculas de ARN que se generan a partir de estas regiones, denominadas crRNA, contiene secuencias de un espaciador y de las repeticiones adyacentes. Los crRNAs actúan como guías para determinadas proteínas con actividad endonucleasa asociadas a CRISPR, denominadas genéricamente Cas, permitiéndoles localizar secuencias diana (también denominadas protoespaciadores), complementarias a la del espaciador de la guía, donde cortarán de forma específica tras reconocer una secuencia adyacente al protoespaciador denominada PAM (por el inglés Protospacer Adjacent Motif). Para poder interaccionar con la secuencia diana, algunas variantes de sistemas CRISPR-Cas (pertenecientes al tipo II, asociados a la endonucleasa Cas9) requieren además del crRNA otra molécula de ARN denominada tracrRNA.
La tecnología CRISPR-Cas, que utiliza componentes de estos sistemas, se emplea para multitud de aplicaciones relacionadas con la interacción programable mediante ARN guías de las proteínas Cas con ácidos nucleicos, ADN y/o ARN dependiendo del sistema concreto. Algunas de estas herramientas están siendo utilizadas desde 2012 para edición genética (es decir, modificación in vivo de secuencias específicas de ADN).
De acuerdo con los estudios de la consultora Grand View Research, el valor del mercado global de la edición genética en 2020 fue de 3700 millones de dólares y estuvo dominado por la tecnología CRISPR-Cas, que supuso el 40,2% del volumen de negocios. Esta misma consultora estima que, a escala global, la cifra de negocios de la tecnología CRISPR-Cas alcanzará los 9600 millones de dólares en 2030.
Entre todas estas herramientas, destacan las basadas en una proteína Cas9 de la bacteria Streptococcus pyogenes (SpCas9). Esta proteína requiere una secuencia PAM excepcionalmente corta (2 pb), lo cual supone una gran ventaja respecto a otras Cas9. Sin embargo, el gran tamaño de SpCas9 supone una limitación para su aplicación in vivo, en particular para la transferencia de la herramienta a células eucariotas. Por este motivo, resulta de gran interés la identificación y caracterización bioquímica y funcional de proteínas Cas9 alternativas, de menor tamaño.
La presente invención está orientada a resolver esta limitación mediante una nueva proteína Cas9 de menor tamaño, que ha demostrado ser apta para su uso en diversas herramientas de biología molecular para modificación genética en procariotas y eucariotas, equivalentes a las implementadas con otras endonucleasas Cas, así como para la producción de antimicrobianos específicos de secuencia.
La presente invención consiste en una herramienta de biología molecular derivada de un nuevo sistema CRISPR-Cas, al que se ha denominado CRISPR-EHCas9 (Figura 1), y su uso en edición genética de bacterias y células de mamíferos. También tiene aplicación como antimicrobiano específico de secuencia y para la digestión programables de moléculas de ADN in vitro.
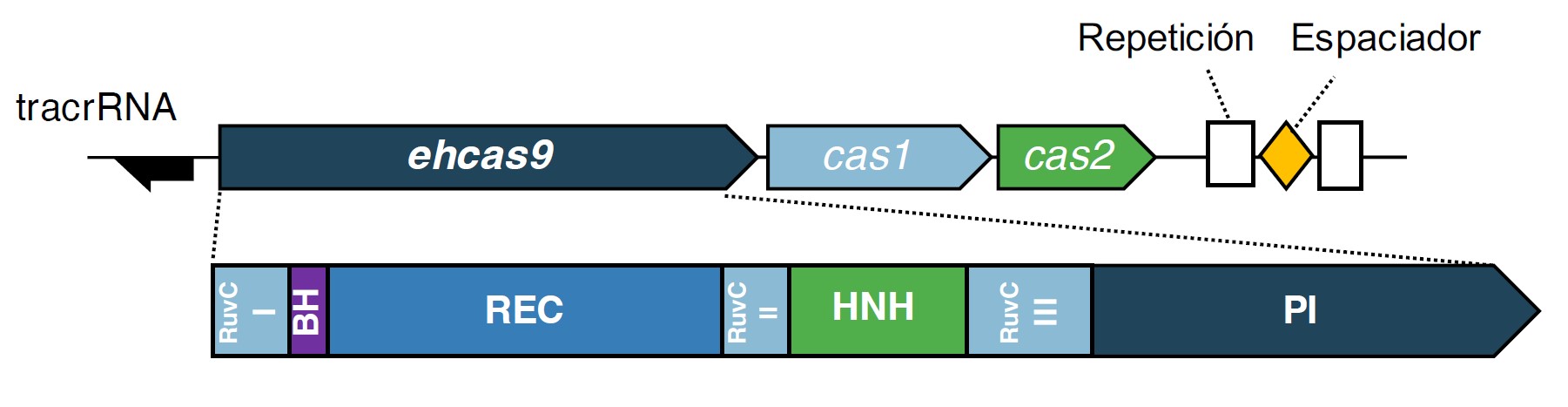
Figura 1. Representación esquemática del locus CRISPR-EHCas9 y los dominios de la proteína EHCas9. El locus CRISPR-EHCas9 comprende tres genes cas en el orden cas9 (denominado ehcas9) - cas1 - cas2 y 2 unidades CRISPR de 36 pares de bases (pb) separados por un espaciador de 29 pb. La ubicación de un posible gen tracrRNA se representa como una flecha que apunta en el sentido de la transcripción. Los dominios de la proteína EHCas9 (representados debajo del locus), incluyen los dominios RuvC (motivos I, II y III), Bridge Helix (BH), de reconocimiento (REC), nucleasa HNH, Phosphate Lock Loop (PLL), WED y de interacción con PAM (PI).
La herramienta de biología molecular CRISPR-EHCas9 comprende:
• La proteína EHCas9, con capacidad para romper moléculas de ADN de forma controlada. Esta proteína tiene un tamaño que facilita su administración tanto a bacterias como células de mamíferos para su edición génica, mediante vectores comúnmente empleados en biotecnología y biomedicina.
• Un ARN guía sintético (EH sgRNA), constituido por una región constante, cuya secuencia es resultado de la combinación de fragmentos del tracrRNA (secuencia en color azul en la Figura 2) y de la repetición CRISPR (secuencia en color naranja en la Figura 2), con una región espaciadora variable de 23 nt, que actúa como secuencia guía y que puede modificarse para que concuerde con la de la diana.
• Los vectores de expresión construidos para la administración a células de los genes que codifican estos elementos y para la producción de dichos elementos. En los diferentes vectores de la invención, la secuencia del gen ehcas9 ha sido optimizada para su expresión en Escherichia coli y para su expresión en células de mamíferos.
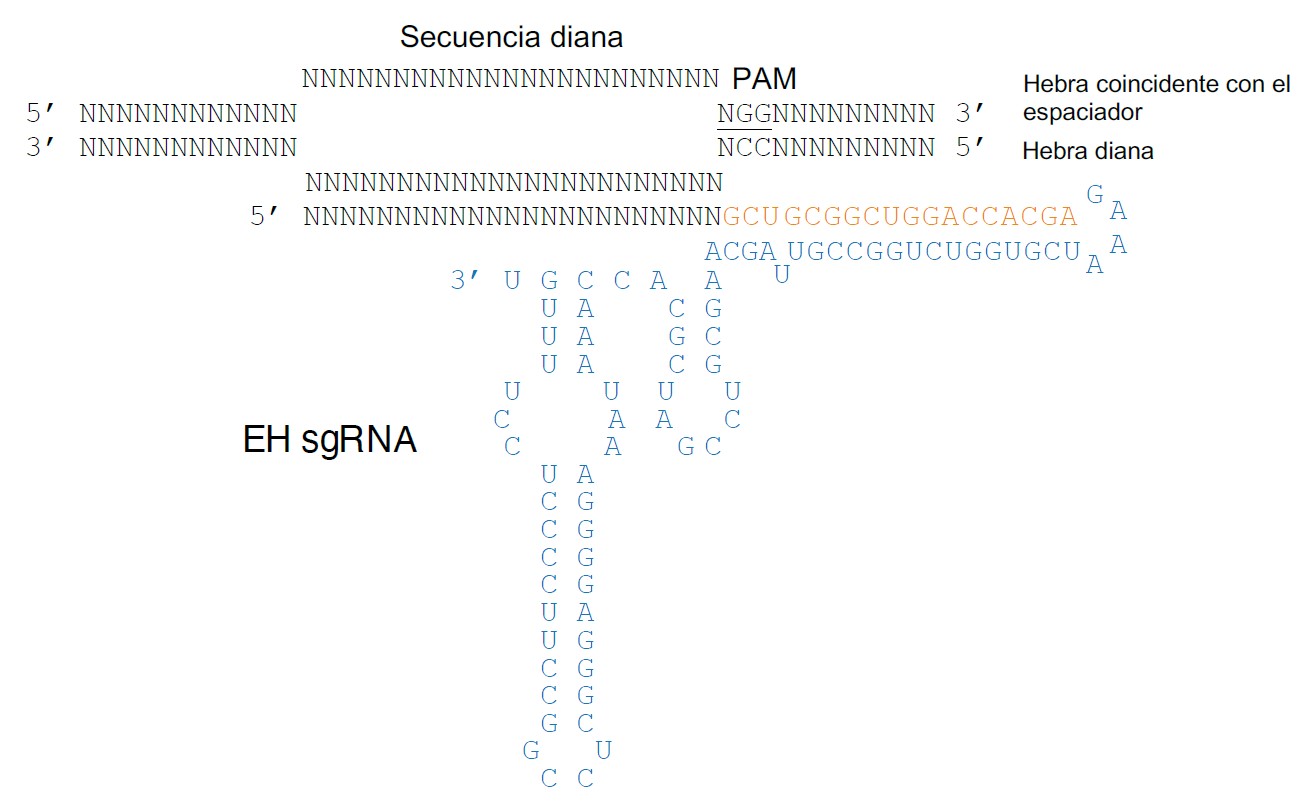
Figura 2. Representación esquemática del complejo formado por un EH sgRNA genérico y una secuencia de ADN diana. EH sgRNA incluye un espaciador genérico de 23 nucleótidos (nt) apareado con la hebra diana en un sustrato de ADN que contiene una secuencia coincidente con el espaciador y un PAM compatible (subrayada). La secuencia de EH tracrRNA se muestra en color azul y la secuencia de la región de repetición en color naranja.
Experimentos in vitro (Figura 3) han demostrado que EHCas9 es una endonucleasa de ADN de doble cadena, específica de secuencia y guiada por ARN, que requiere iones Mg+2 para su actividad (Figura 3A) y corta en ambas cadenas de secuencias diana a una distancia de 3 nt respecto al motivo PAM NGG. Además, EHCas9 exhibe una actividad óptima a temperaturas en torno a los 37ºC, compatibles con la fisiología de células de animales y la de bacterias que interaccionan con estos (Figura 3B). Su excepcionalmente estrecho rango de temperaturas de funcionamiento hace que su actividad sea fácilmente controlable modulando las condiciones de incubación.
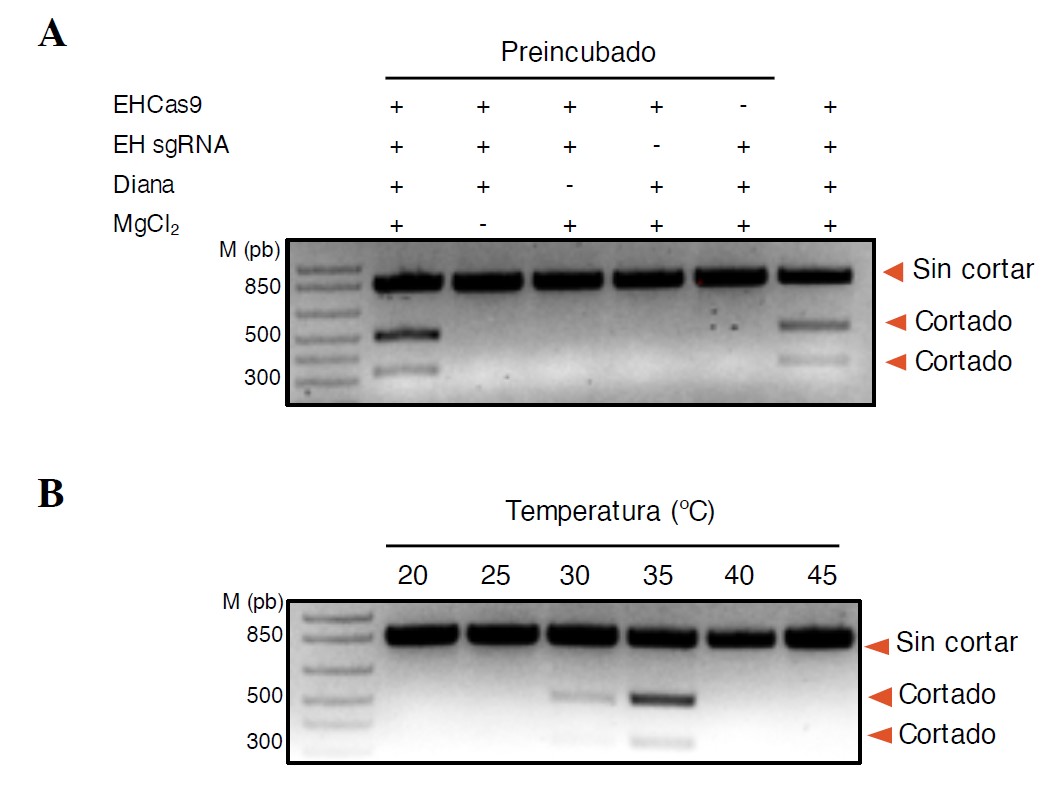
Figura 3. Electroforesis en geles de agarosa de productos de reacción de EHCas9 obtenidos mediante ensayos de digestión in vitro con sustratos de ADN de doble cadena. (A) Muestras de reacciones de digestión en condiciones estándar utilizando todos los componentes de la reacción con el complejo EHCas9:EH sgRNA preincubado (carril 2) o sin preincubar (carril 7), y en ausencia de algún componente (MgCl2, carril 3; diana con PAM, carril 4; EH sgRNA, carril 5; EHCas9, carril 6). (B) Muestras de las reacciones de digestión a diferentes temperaturas de incubación.
La herramienta CRISPR-EHCas9 ha demostrado su capacidad de seleccionar aquellas células de la bacteria Escherichia coli que hayan sufrido mutaciones en experimentos de edición genética, sin la necesidad de tener que introducir marcadores de selección en el genoma del hospedador, simplificando de esta manera el procedimiento de obtención de mutantes delecionados de secuencias específicas. La Figura 4 muestra los resultados obtenidos en experimentos de edición del gen pyrF (GDI). Las colonias provienen de la co-transformación de un molde de recombinación (la recombinación daría lugar a una deleción de 0,6 kpb en pyrF), y un plásmido que codifica para EHCas9 y un EH sgRNA dirigido a una secuencia diana en el gen pyrF (+EHCas9) o con un plásmido equivalente pero que solo codifica el EH sgRNA (-EHCas9). Los resultados muestran que, cuando se dirige al cromosoma de E. coli, la herramienta CRISPR-EHCas9 provoca muerte celular, demostrando así su eficacia como agente antibacteriano específico de secuencia.
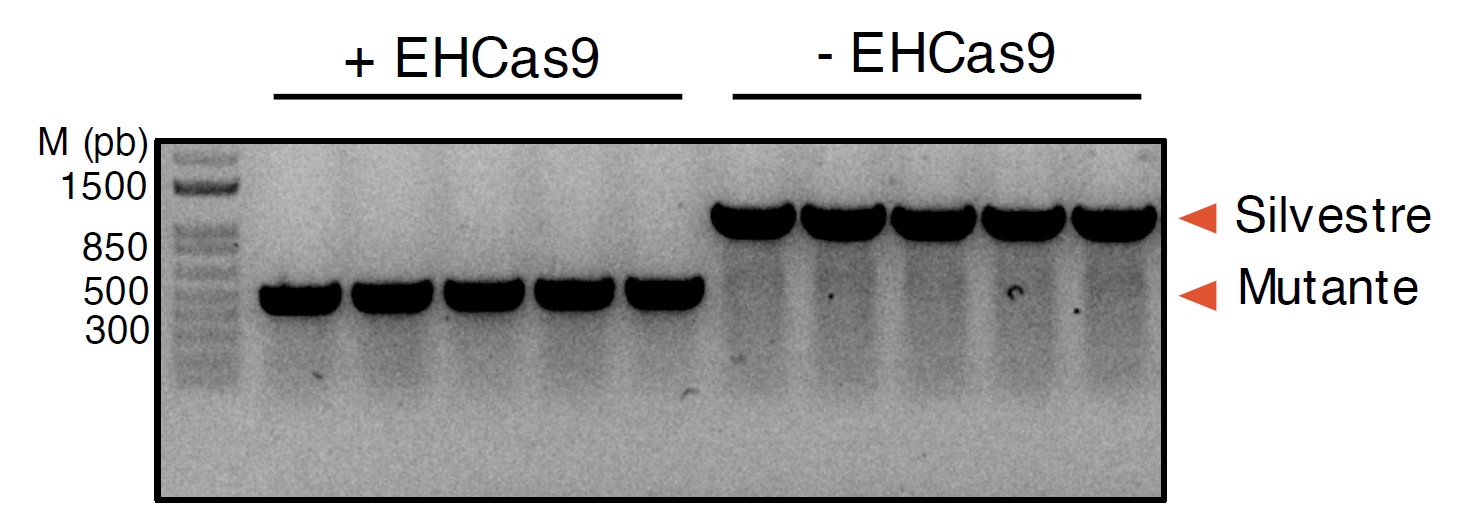
Figura 4. Edición genética de E. coli asistida por EHCas9. Electroforesis en gel de agarosa de productos de PCR obtenidos a partir de colonias de transformantes obtenidas en experimentos de edición del gen pyrF (GDI). Cada carril se corresponde con una colonia elegida al azar. Se señala las posiciones esperadas para las bandas correspondientes al gen pyrF original (Silvestre, ca. 1 kpb; WT) y el del gen con la deleción (Mutante, ca. 0,5 kpb).
Por último, se ha comprobado que la herramienta CRISPR-EHCas9 también facilita la edición genética de células N2a de ratón, provocando deleciones en la región de la secuencia diana (Figura 5). Si bien la eficiencia de edición por SpCas9 es superior a la obtenida con EHCas9, la proteína de la invención mostró una menor tolerancia a variaciones en la secuencia PAM, lo que se traduce en una mayor especificidad de la edición. Por otra parte, es de destacar el hecho de que los ensayos de toxicidad celular realizados con la herramienta de la invención no muestran diferencias significativas con el comportamiento mostrado por SpCas9.
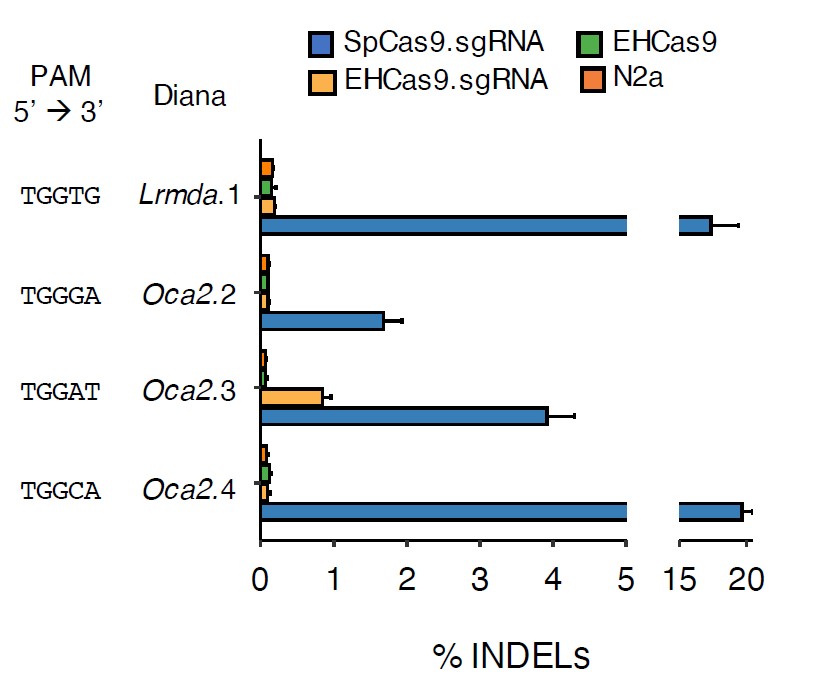
Figura 5. Edición genética mediada por EHCas9 en células N2a de ratón. Porcentaje del número de lecturas de secuenciación con inserciones o deleciones obtenidos para 4 secuencias diana del genoma de ratón tras la transfección con plásmidos que codifican para SpCas9 y Sp sgRNA (SpCas9.sgRNA; azul), EHCas9 y EH sgRNA (EHCas9.sgRNA; naranja) o EHCas9 (EHCas9; verde). Como control negativo se incluyen los resultados obtenidos con células no transfectadas (N2a; rojo). A la izquierda, se muestran las secuencias PAM de cada diana.
Es de esperar que la herramienta CRISPR-EHCas9 también sea funcional en otros procariotas y eucariotas, tanto para estas como para otras aplicaciones basadas en el reconocimiento y posterior modificación de secuencias específicas de ADN, tal y como se ha descrito previamente para otras herramientas basadas en Cas9 nativas y sintéticas derivadas de estas. Estas aplicaciones también incluyen el diagnóstico molecular.
En resumen, la invención se refiere al uso de la proteína EHCas9, de la secuencia de ADN que la codifica, del vector de expresión construido para su administración a células, de la célula que contiene dicho vector, y/o del sistema CRISPR-EHCas9 de la presente invención para:
• la modificación genética, regulación de expresión génica y/o visualización in vivo de secuencias nucleotídicas concretas; y/o
• el diagnóstico molecular de enfermedades; y/o
• la producción de antimicrobianos específicos de secuencia.
VENTAJAS DE LA TECNOLOGÍA
La tecnología CRISPR-Cas presenta ventajas con respecto a las tecnologías de modificación genética empleadas de forma convencional en sectores como el agroalimentario, como es el caso del empleo de sustancias químicas con acción mutagénica o de radiaciones ionizantes, entre las que se destaca:
• Direccionamiento a genes específicos
• Ausencia o reducción de modificaciones indeseadas
Con respecto a otras metodologías de edición genética dirigida, como las basadas en nucleasas efectoras de tipo activador de transcripción (TALEN, del inglés transcription activator-like effector nuclease), nucleasas de dedos de zinc (ZFN, del inglés zinc-finger nucleases), o la mutagénesis dirigida por oligonucleótidos (ODM), la herramienta presenta las siguientes ventajas:
• Facilidad de uso
• Menor coste
Además, la herramienta de esta invención presenta las siguientes ventajas con respecto a otros sistemas CRISPR-Cas9:
• Menor tamaño de la proteína EHCas9 (alrededor del 78% del tamaño de SpCas9).
• Mayor facilidad de administración tanto a bacterias como a células de mamíferos.
• Permite la incorporación, en una única molécula vector, de secuencias de elementos genéticos accesorios, como secuencias reguladoras o moldes para la edición genética.
• Facilita la administración de derivados inactivos de la nucleasa fusionados con péptidos con distintas actividades relacionadas con el ADN.
• Rango de temperaturas de funcionamiento excepcionalmente estrecho, lo que permite controlar fácilmente su actividad modulando las condiciones de incubación.
• Requiere la presencia de un motivo PAM muy corto y frecuente, lo que facilita la selección de secuencias diana.
• La menor tolerancia a variaciones en la PAM da como resultado una mayor especificidad en relación con SpCas9.
• Puede ser empleada como un sistema muy eficaz de selección positiva de bacterias mutantes, sin necesidad de introducir marcadores de selección.
ASPECTOS INNOVADORES DE LA TECNOLOGÍA
El sistema CRISPR-EHCas9 ha sido identificado por investigadores del grupo de Microbiología Molecular de la Universidad de Alicante en un metagenoma generado a partir de una muestra de agua recolectada en el Parque Natural El Hondo (España), un entorno natural previamente inexplorado.
La comparación de la secuencia de aminoácidos de la proteína EHCas9 con las de las proteínas Cas9 disponibles en bases de datos muestra una identidad de secuencia inferior al 68%. Cuando la comparación se realiza frente a proteínas nativas Cas9 empleadas en la edición de genomas en células de mamíferos, se observa una identidad de secuencia inferior al 29%. Además, la secuencia de interacción con PAM difiere considerablemente.
A diferencia de las proteínas Cas9 disponibles en la actualidad, el sistema de esta invención permite que se puedan incorporar, en una única molécula vector, secuencias de elementos genéticos accesorios, como secuencias reguladoras o moldes para la edición genética.
La tecnología está evaluada y testada en laboratorio. Se ha demostrado que la nueva herramienta CRISPR-EHCas9 es funcional in vitro para la restricción programable de secuencias de ADN de doble cadena y para aplicaciones de edición de genomas tanto en un procariota (bacteria Escherichia coli) como en un tipo celular de un eucariota (células N2a de ratón). Además, ha demostrado su eficacia como agente antibacteriano específico de secuencia.
La presente invención se enmarca en el campo de la ingeniería genética. Más concretamente, el objeto de la invención se refiere a nueva proteína endonucleasa EHCas9 y a un sistema CRISPR-EHCas que comprende dicha proteína para la edición genética en células y producción de antibacterianos.
Los sistemas CRISPR-Cas permiten el silenciamiento o la eliminación de genes, mutagénesis, y correcciones de secuencias específicas del genoma celular de una manera fácil, rápida, y altamente precisa. Entre sus numerosas aplicaciones destacan el diagnóstico y tratamiento de enfermedades, así como la producción de antimicrobianos específicos de secuencia.
La Unión Europea está valorando excluir de la legislación sobre OMG los vegetales producidos mediante nuevas técnicas genómicas. De ser así, el uso de los sistemas CRISPR-Cas en el sector agroalimentario podría verse potenciado en el mercado europeo.
Por lo tanto, la herramienta de esta invención tendría aplicación en empresas agroalimentarias, biotecnológicas, medioambientales, bioquímicas y de biología molecular, y del sector la salud.
Se buscan empresas interesadas en adquirir esta tecnología para su explotación comercial mediante:
• Acuerdos de licencia de la patente.
• Acuerdos de cooperación técnica, a través del desarrollo de proyectos de I+D conjuntos o asistencia técnica personalizada, para adaptar la tecnología a las necesidades de la empresa o desarrollar nuevas aplicaciones o herramientas.
• Acuerdos de subcontratación para asistencia técnica, formación, etc.
Perfil de empresa buscado:
• Empresas del sector biotecnológico
• Empresas del sector biomédico
• Empresas del sector farmacéutico
• Empresas del sector agroalimentario
La presente invención se encuentra protegida mediante solicitud de patente nacional:
• Título: “Proteína endonucleasa Cas9 y sistema CRISPR-Cas asociado”.
• Número de solicitud: P202230911.
• Fecha de solicitud: 21 de octubre de 2022.
Biología Molecular y Biotecnología
Medicina y Salud

Carretera de Sant Vicent del Raspeig s/n - 03690 Sant Vicent del Raspeig - Alacant
Tel.: 965 90 9959




